守屋、北野プロジェクトに行く
まずは、遺伝子つなひき法の誕生からである。それは、守屋が2004年に北野宏明氏が統括を務めるERATO-SORST北野共生プロジェクトの研究員として研究を始めたところから始まる。守屋は計算機の力、特にコンピュータシミュレーションを使って生物を理解しようというシステム生物学に憧れていた。そのあたりは守屋のHPの私にとっての「システムバイオロジー」と「私がシステムバイオロジーを選んだのはなぜか?」に書いた。セントルイスでの第4回ICSBで守屋は北野プロジェクトに参加することが決まったのだが、その時にロバストネスの概念を初めて聞いた。「細胞内パラメータをどれだけ変動させられるか?」というのがロバストネスの理解の鍵だということを知った。今までそんなものが重要な視点になるとは思っていなかった守屋は、直感的にこれは間違いなく新しい概念であると感じた。さらに、当時のロバストネスの研究はほとんどシミュレーションを通じた理論的なものに限られており、それを調べるという実験手段が存在しなかった。守屋は自身が培ってきた酵母の分子遺伝学の技能を使えば、それが調べられると感じた。つまり、誰もやったことがないことが自分にはできると思ったのだ。
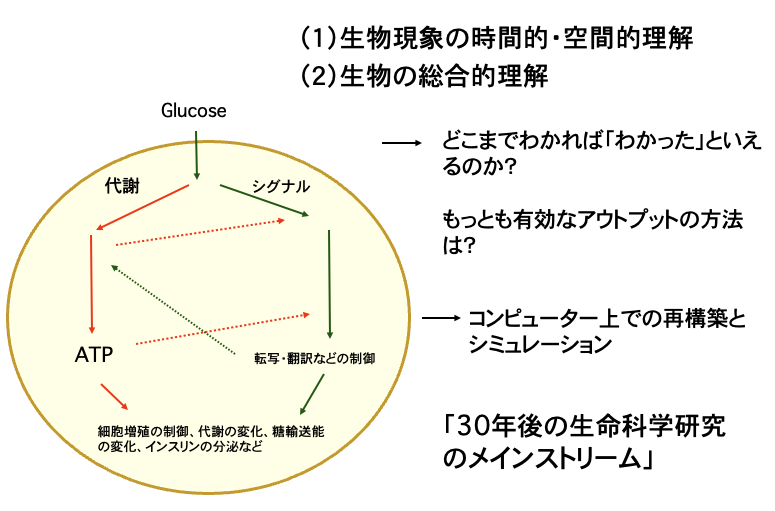

北野プロジェクトに着任、gTOWのアイデアが生まれるまで
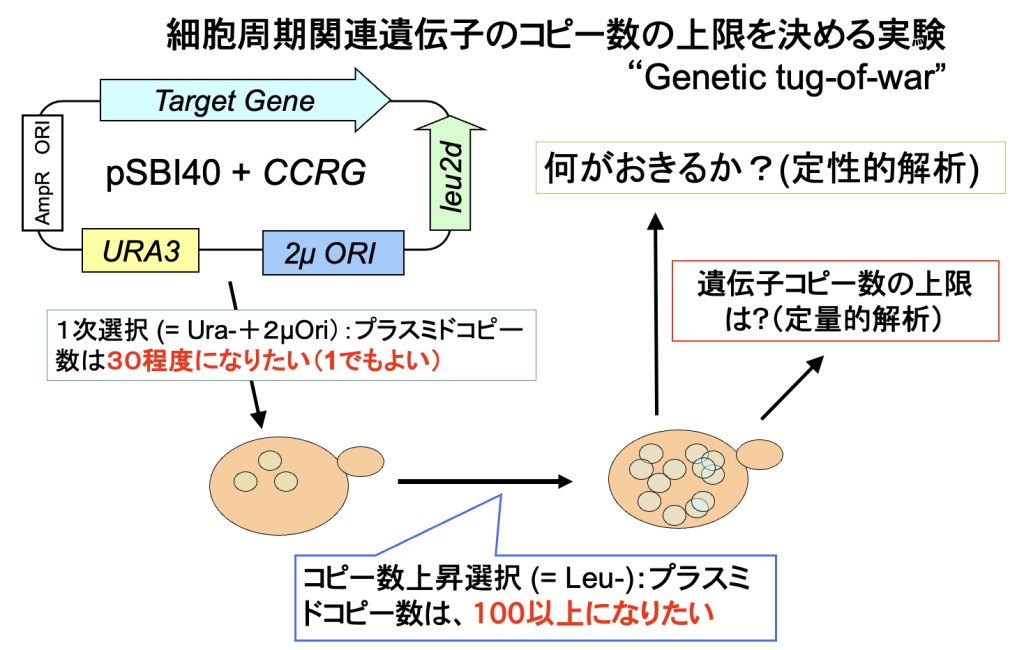
北野プロジェクトに着任した直後は、半年ほど生物学実験室がなかった。守屋はその間、原宿の表参道に面したオフィスで、シミュレーションでロバストネスがどのように現れてくるかを研究していた。それと同時に、どうすればロバストネスを実験で調べられるかを考えていた。見たいことは、「細胞機能に悪影響が出る/出ない細胞内パラメータの範囲はどれくらいなのか?」ということである。細胞機能とは細胞周期でありその異常はDNA含量で調べられる。パラメターは遺伝子発現量とした。6月の北野氏のディスカッションで、実験のコンセプトが初めて出てくる。そこでは、多コピープラスミドで遺伝子コピー数をばらつかせ、その時の発現量(GFP融合タンパク質)を検出し、その結果起きる細胞周期の異常をDNA含量として検出する。これをフローサイトメトリー(FACS)か蛍光顕微鏡での細胞集団の分布として検出するというものだった(下図)。
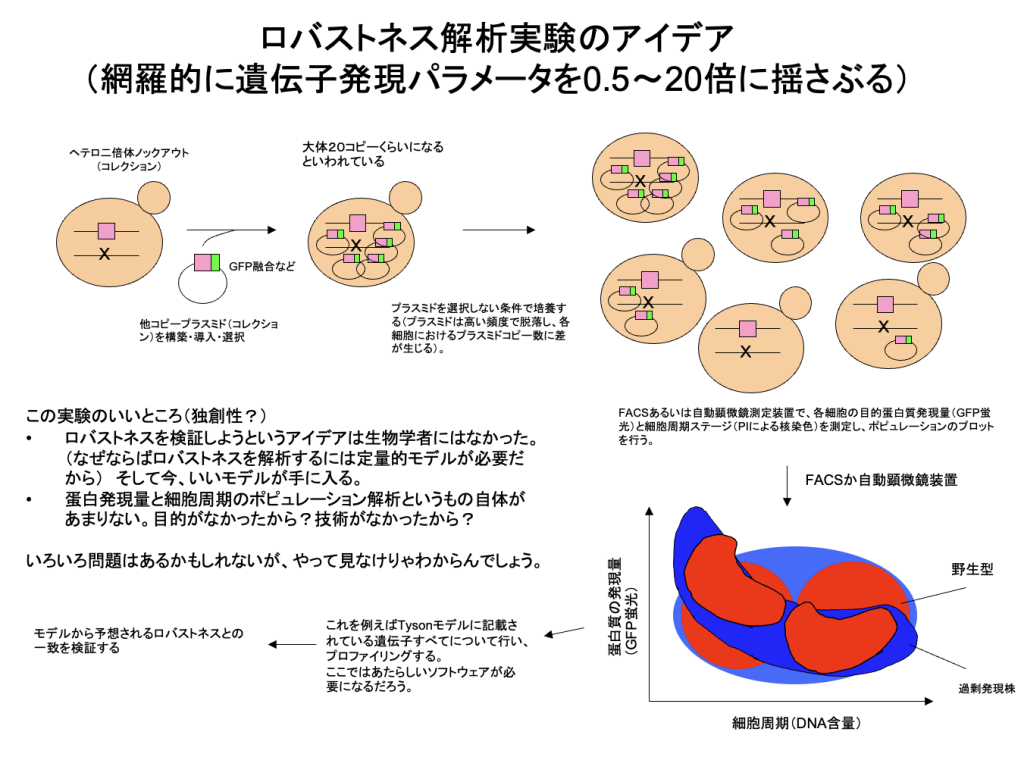
これはこれでパラメータ変動と表現型を一網打尽にできるアイデアだし、いまでも(今では)実行可能かも知れない。実際、GFP融合によるタンパク質発現量取得は、最新のgTOW系(2.0)で行っている。しかし、当時はまだFACSや蛍光顕微鏡で細胞周期の異常表現型が網羅的に取得できるのか確信がなかった。
この後、いくつかの変遷があった。GAL1pro-CENを使ってプラスミドのコピー数をばらつかせた後固定させるということも考えていた。
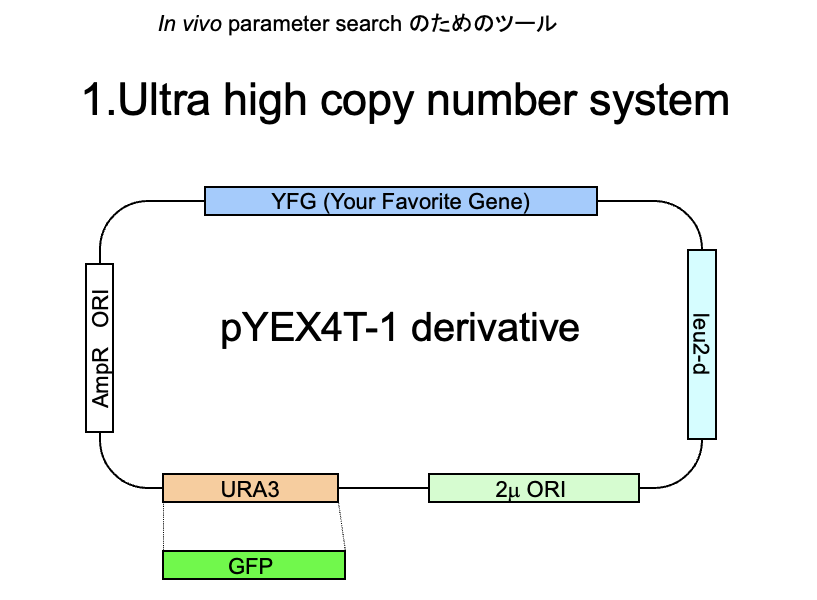
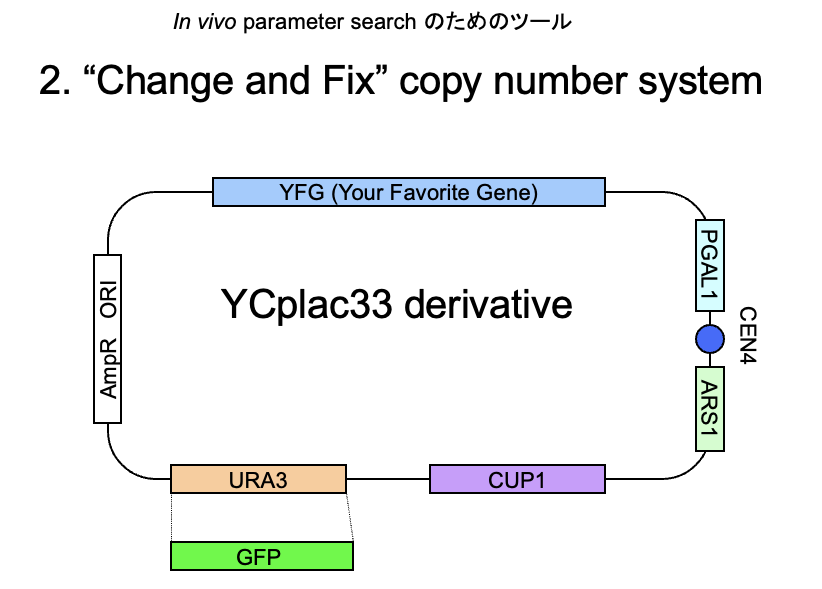
2004年10月の北野氏とのディスカッション資料(2/3)
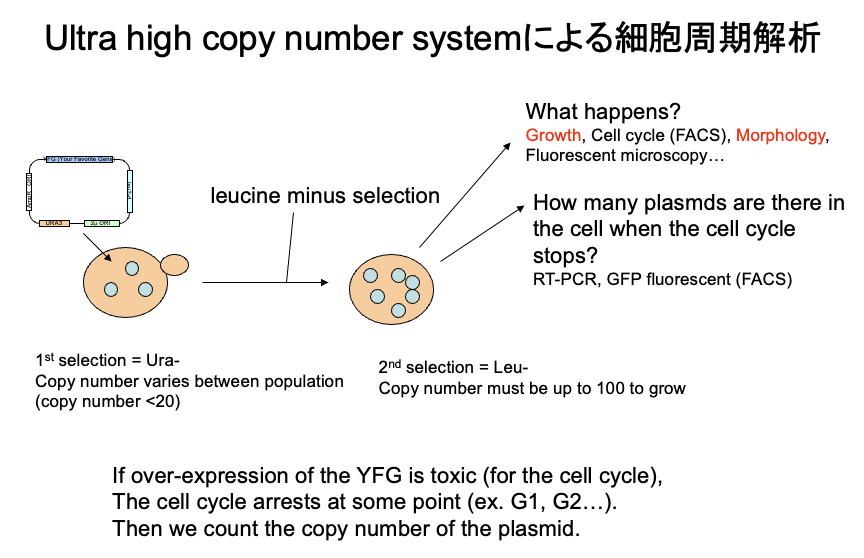
2004年10月の北野氏とのディスカッション資料(3/3)。現在のgTOWのアイデアと同じ。名前はまだついていない。たしか、系の頭文字をとってUHICONとか言ってた記憶がある。
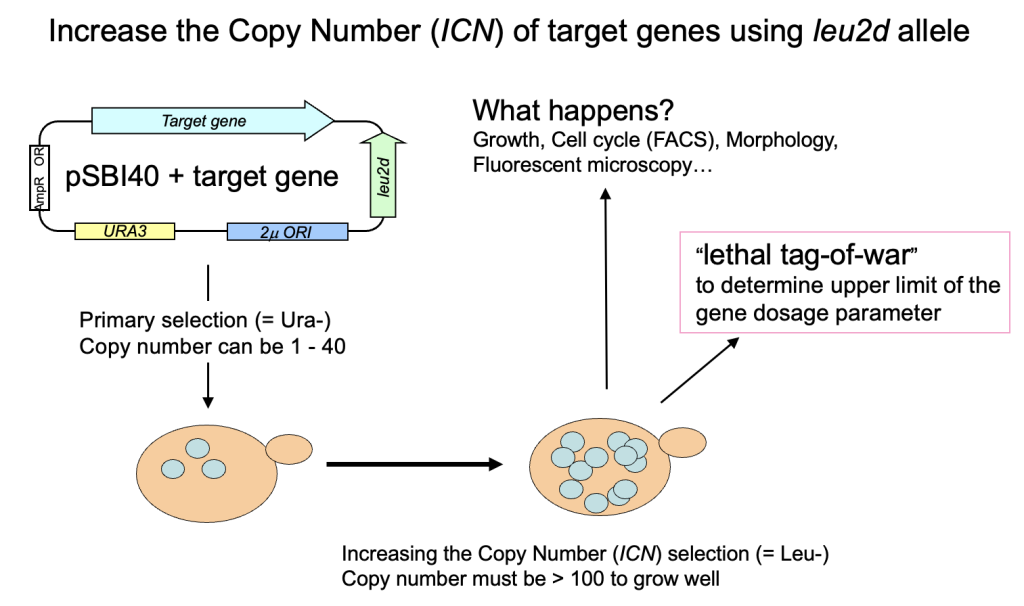
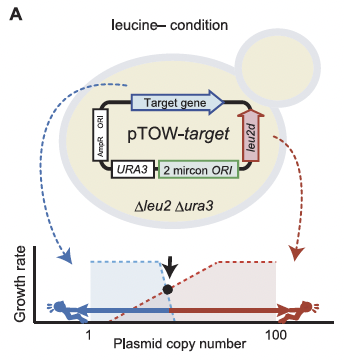
その後、実験室が完成し、2004年9月29日の実験ノートの最初のページでは、すでに現在のgTOW実験のためのプラスミド構築が始まっている。遺伝子つなひき法についても守屋のHP(遺伝子つなひき法)に詳しく書いてある。gTOWの(上記の実験系にない)最も重要なポイントは、標的遺伝子の限界発現になるまでプラスミドコピー数を上げられる系だということ、さらにプラスミドコピー数を測れば、もともとの発現量から何倍増やしたら増殖阻害が起きるかが分かるというところにある。つまり、欲しかった「細胞機能に悪影響が出る遺伝子発現量が測れる」実験系なのだ。しかも、やることは簡単で、標的遺伝子が入ったgTOWプラスミドを作って酵母にいれ、条件を変えて培養するだけ。酵母が勝手に限界を決めてくれる。
この系が動いているかは増殖速度を見ればわかる。これを多数の標的で行うためにマイクロプレートリーダーを用いた培養・増殖測定系を作った。プラスミドコピー数を測るためにリアルタイムPCRの実験系を作った。マイクロプレートリーダーを用いた増殖測定(+蛍光測定)は、現在でも守屋研のもっとも強力な実験手法の1つとなっている。リアルタイムPCRはなかなかトリッキーで使える人が限られているのと、コピー数測定が必須ではないためあまり使われていないが、できれば使いたいところではある。
「genetic Tug-Of-War」という名前がつくまで
さて、gTOW、すなわちgenetic Tug-Of-Warという名前はいつついたのだろうか?はじめに登場するのは、2005年4月の「プロジェクト説明2」というプレゼンである。これが何のプレゼンだったのかは分からないのだが、ここで「lethal tag-of-war」が出てくる。このプレゼンの時に一緒にいたイギリス人の同僚に、「つなひき」の綴りが違うといわれたのを憶えている。tug-of-warをtag-fo-warと間違って書いている。いずれにせよ、この頃までには30の細胞周期の遺伝子の解析結果が出始めていて、外に向けたプレゼンテーションをあれこれ考えているうちに、この説明の方法に行きついたのだろう。今にも繋がるつなひきの図も描かれている。
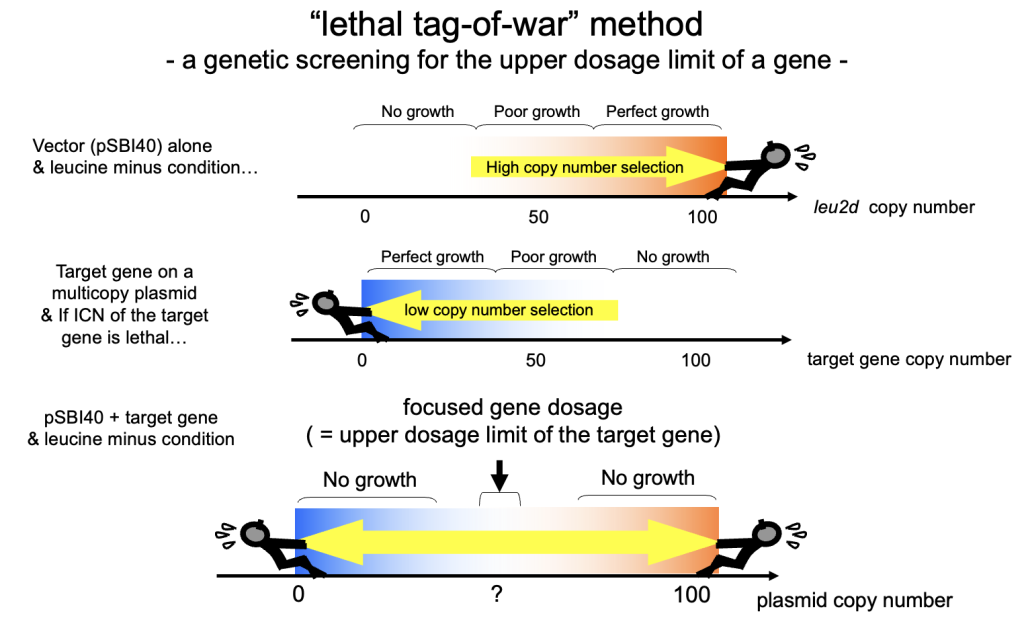
そして、2005年の7月の酵母細胞研究会で、「Genetic tug-of-war」がいよいよ登場する。
Chen2004モデルとFred Cross
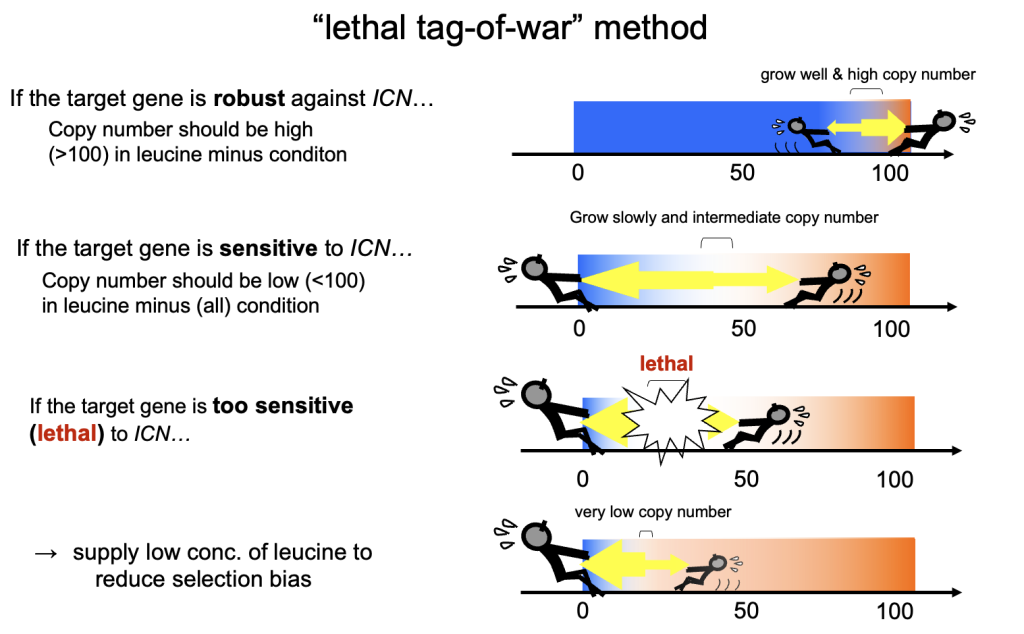
ここで少し脇道にそれる。gTOWの意義を高めた細胞周期の数理モデルと天才Fred Cross氏について語る。gTOWでは、標的の遺伝子の発現が元のレベルから何倍の限界を持っているか決める事ができる。この限界が何倍だったら、細胞システムはロバストといえるのだろうか? また、ロバストだったとして(あるいはロバストではなかったとして)、それはどういう原理でそうなっているのだろうか? まず前者だが、そもそも誰もちゃと測ったことがないので、その評価軸は存在しない。当時の私は、ほとんどの遺伝子について100倍以上は大丈夫なんじゃないかとなんとなく思っていた。ただ、測ってみるといろいろで、少ないものは2倍以下だった。私にとってはすごい驚きだったが、みんながそう思うとは限らない。後者について、ロバストネスとはシステムが複雑に働いて作り出される性質なので、そもそも直感的に説明できるものでもない。いずれにせよ、ロバストネスを比較するための土台のようなものが必要だった。それが、数理モデルだという事になる。
そして、幸か不幸か、私が北野プロジェクトに着任したその年に、バージニア工科大学のJohn Tysonらのグループから出芽酵母細胞周期の統合的な数理モデルが発表されたのだ(Chen MBoC 2004)。当時彼らはこれをCyber Yeastと呼んでおりHP上でシミュレーションもできた。いずれにせよ、このモデルと自分たちの実験結果を比較し、ロバストネスが理論的にどれだけ予測できるのか/できないのかが評価できる。そういうわけで守屋はこのモデルに組み込まれている30種類の細胞周期遺伝子をgTOWの最初のターゲットにしたのだった。ところが、このモデルの論文の著者には1人とんでもないくせ者、否、細胞周期業界の大御所がいた。それがFred Cross氏である。彼は実験家にして自らシミュレーションもできる。実際、この論文で「Robustness assay」として、パラメータを変動させたときの細胞周期のロバストネスをシミュレーションで調査している(Cross氏が作ったMATLABのスクリプトは、その後Cross氏から頂いて自身でロバストネス解析を行えるようになった)。
問題はその後だった。何かのついでに東京を訪れたFred Cross氏が、北野プロジェクトを訪問してセミナーをするということになったのだ。当時の北野プロジェクトは原宿にあったこともあり、国内外からたくさんの研究者が訪れていた。「XXXさんが来るから集まって」といわれて、突然英語のプレゼンをする流れになったこともある。まあそれは良いとして、そこでCross氏の発表の要旨を見たときにビビった。「ロバストネス解析を実験でやった」と書いてあったからだ。「やりたこと先にやられた!」と守屋はショックを受け、北野さんにそれを言ったが、「まあ同じ事はやっていないだろう」、ということでとりあえず気にしないことにした。実際、コンセプトは一緒だがやり方は違っていた(Cross MBoC 2005)。そしてその後、Fred Cross氏は細胞周期のシステムバイオロジーにおいてたて続けにすごい論文を発表された。そもそも戦って勝てる相手ではなかったのだ。
gTOW論文発表とさきがけ「生命システム」への採択
さて、話を戻そう。そんなこんなで(?)、gTOWの最初の論文が発表となる。この論文の作成においてもいろんな事があったが、その辺は割愛する(論文書くときの苦労話は、昔のブログをまとめたものを読んで欲しい)。ちなみに、2006年4月に投稿し2006年7月に出版されたこの論文で、gTOWの図が完成した。
さて、ちょうどこの頃、JSTさきがけに「生命システム動作原理と基盤技術」という、システムバイオロジーを推進する研究領域が立ち上がった。ロバストネス解析は、まさにシステムバイオロジーを代表する研究テーマだ。守屋はこれに採択されることに心血を注いだ。その時の面接は、終わってから何日もフラッシュバックするほど自分にとってはすさまじいプレッシャーだった。以下は、申請書の抜粋。
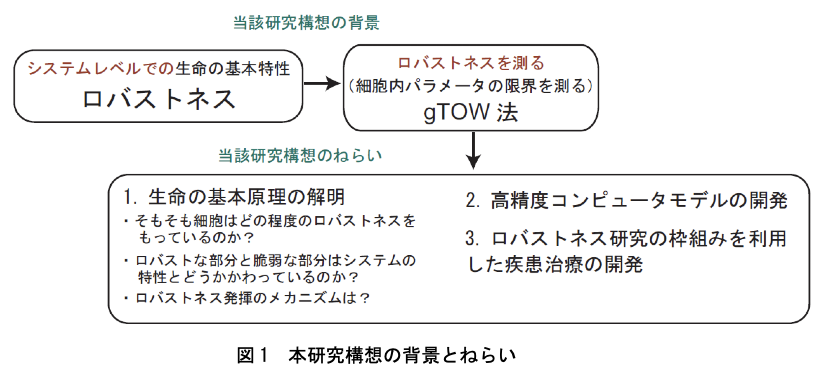
Ⅱ.研究のインパクトと将来展望 本研究構想の達成により与えられる最大のインパクトは、これまでの生物学研究で全く対象とされてこなかった「細胞内パラメータの許容範囲」という量的データを実験的に取得すること(すなわち細胞のロバストネスを実験的に測定すること)が、いかに生命現象の理解に貢献するかを実証できることにあると考える。ロバストネスはシステムレベルで生命をとらえたときに始めて理解が可能になる生命の“システムとしての”特徴である。従って言い換えると、本研究構想の達成による最大のインパクトは「生命のシステムレベルでの研究を特徴づけるような研究アプローチはどのようなものなのか?」さらに、「そのシステムレベルでの研究アプローチによってどのようなことがわかり、それがいかに生命現象の基本的な原理の理解に(そしてさらに医療への応用に)重大な貢献ができるか?」ということを実証することができるところにあると考える。
さきがけ生命システムに採用されたことは、gTOWにとって最も意味のあることだった。そもそも、北野氏以外にgTOWの潜在性を認められたことがとても大きい。さらに、この領域でもまれなければ、gTOWのその後の展開はなかっただろう。実際、「遺伝子綱引き法」という日本語名は、このさきがけの研究報告会のために考えたものだった。当時は、「綱引き」を漢字で書いた。現在はひらがなになっている。
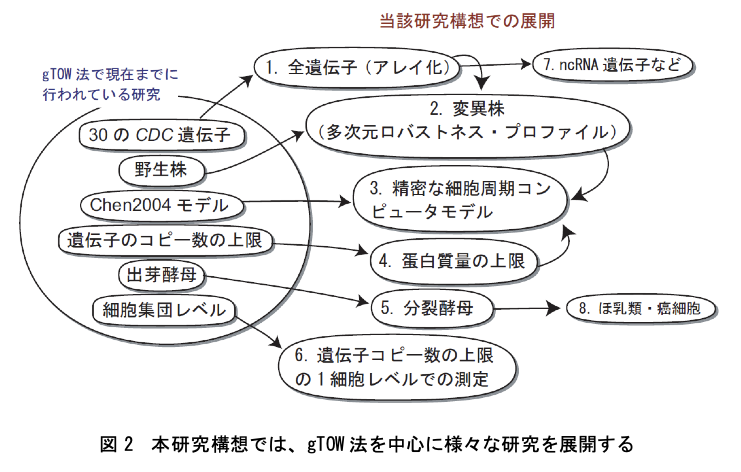
ちなみに、gTOWについては、それを説明するためのページを作ったし(遺伝子つなひき法)、いくつかの日本語総説も書いた。2007年の実験医学の解説記事や2009年の現代化学の解説記事で、これは当時ブログやプレゼンのために作った内容を全部盛りこんだものになっている。2006年10月から始まったさきがけ研究は、2009年2月までは、癌研究所のシステムバイオロジー部という、北野プロジェクトが借りている部屋で行われていた。出芽酵母の細胞周期のgTOW解析に続き、分裂酵母の細胞周期のgTOW解析、さらに出芽酵母のゲノムワイドなgTOW解析が北野プロジェクトの共同研究として進んでいた(これらは第2部で説明する)。
最も初期のgTOW論文を支えてくれたのは当時北野プロジェクト/ソニーCSLの吉田由紀氏だった。特にMoriya PLoS Genet. 2006ではプラスミドコピー数とタンパク質発現量の関係を調べるためにウエスタンブロッティングをやりまくったが、それらはすべて吉田氏がやってくださった。吉田氏は守屋がポスドクをした三菱化学生命科学研究所の酒井明氏の研究室で助手をされていて、守屋と入れ替わる形で退職されたという不思議な縁である。私は吉田さんの仕事を引き継いで論文にしたので、そこでも共著論文がある(Moriya, Yoshida, et al, Genes Dev. 2001)。